
本制品是基于CRISPR/Cas9基因编辑技术而研发的sgRNA (single guide RNA)体外转录合成试剂盒。使用本试剂盒,用户仅需按照本试剂盒说明书设计并合成Target-specific DNA oligo,就可以通过本试剂盒每次反应获得10~40μg sgRNA。获得的sgRNA可以用于体外分析和鉴定sgRNA的效果,也可以用于转染表达Cas9的细胞以实现目的基因的基因编辑。
本试剂盒通过PCR扩增和依赖于T7 RNA聚合酶的转录两步法高效产生靶向目的基因的sgRNA。本试剂盒提供了预混合的Scaffold Template,Scaffold Template包括与使用者设计的目标特异性序列部分重叠的长度为80nt序列以及用于扩增该序列的引物,退火形成互补链后由DNA聚合酶进行聚合,并最终生成用于转录的双链DNA模板。利用本试剂盒提供的试剂和使用者自己设计的特异性DNA序列,单管反应可在0.5~4h内获得10~40μg功能性的sgRNA。纯化后的sgRNA可以转染细胞,在表达Cas9的细胞中进行基因编辑,也可以与Cas9核酸酶混合进行酶切鉴定实验。
一步法sgRNA合成试剂盒(货号:YTB4152)和两步法sgRNA合成试剂盒(货号:YTB4151)的主要差别在于一步法操作简单仅需一步反应,但产量仅为两步法的约一半;两步法操作略复杂,但每个反应的产量比一步法多约1倍。

sgRNA由18~20bp与靶基因序列互补的CRISPR RNA (crRNA)序列以及能与Cas9特异性结合的trans-activating crRNA (tracrRNA)序列组成。在细胞内,Cas9核酸酶可以特异性地结合sgRNA,同时sgRNA也能特异性地与相应的靶基因序列配对结合,这样就可以导致Cas9核酸酶在靶基因位点的PAM (proto-spacer adjacent motif)序列NGG上游大约三个碱基的位置对靶基因进行切割。随后在细胞DNA修复过程中会导致基因靶位点处的插入、删除或替换,从而可能产生移码突变,导致目的基因的缺失突变。

本试剂盒通过两步法合成sgRNA的原理请参考图1。其中第一步通过PCR反应扩增获得用于RNA转录的模板DNA,第二步通过RNA聚合酶合成sgRNA。
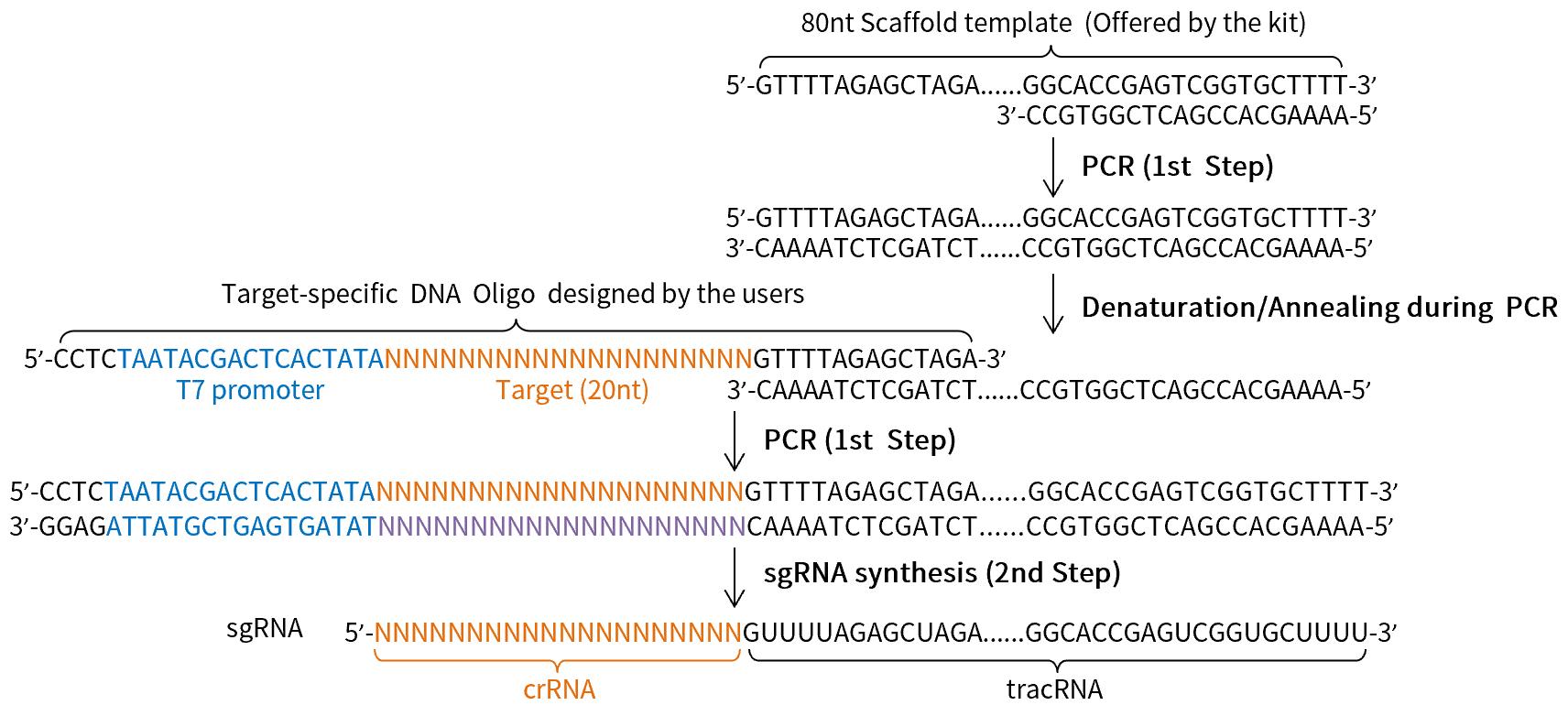
图1.两步法sgRNA合成试剂盒合成sgRNA的原理图。

| 组分 | 20T | 100T |
| 2×Template Mix | 200μL | 1mL |
| BalbFusion DNA聚合酶 | 11μL | 55μL |
| 2×sgRNA Synthesis Buffer | 200μL | 1mL |
| RNase抑制剂(40U/μL) | 20μL | 100μL |
| T7 RNA聚合酶 | 20μL | 100μL |
| DNase I | 40μL | 200μL |
| 超纯水 | 400μL | 2mL |
保存:-20℃,有效期1年。

- 酶使用时宜存放在冰盒内或冰浴上,使用完毕后宜立即放置于-20℃保存。
- 严格使用RNase Free的实验器材(如吸头、离心管、PCR管等),并在操作过程中严格注意避免RNase污染。
- 需要自备的主要试剂:3M NaAc (pH5.2);无水乙醇;70%乙醇;1:1水饱和苯酚/氯仿混合溶液。如需更多的超纯水,推荐选购无菌无酶超纯水。

- 两步法sgRNA合成试剂盒合成sgRNA的效果请参考图2。
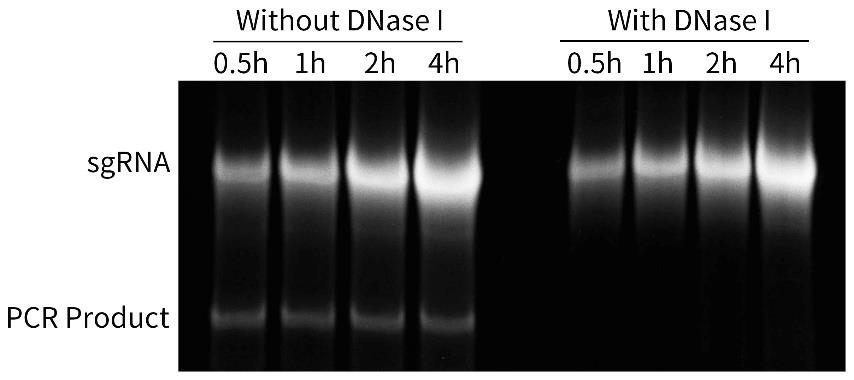
图2.两步法sgRNA合成试剂盒合成sgRNA的效果图。参考本试剂盒的使用说明设置20μL PCR反应体系并进行PCR反应。参考本试剂盒的使用说明,设置20μL转录反应体系,37℃分别孵育0.5h,1h,2h,4h,70℃孵育10min以终止反应。取出5μL反应液,加入1μL 6×DNA上样缓冲液作为模板消化前的样品;另外取出5μL反应液,加入1μL DNase I,37℃孵育15min以消化DNA模板,加入1μL 6×DNA上样缓冲液作为消化模板后的样品。将模板消化前后的样品进行15%非变性聚丙烯酰胺凝胶电泳,电泳结束后使用NadRed红色核酸染料室温染色15min,使用凝胶成像系统观察实验结果。如图所示,使用本试剂盒可以很好地合成sgRNA,并且在图中所示的反应时间内,反应时间越长,合成的sgRNA的产量越高。不同实验条件产生的效果可能有所不同,图中效果仅供参考。 - 两步法sgRNA合成试剂盒合成的sgRNA引导Cas9核酸酶切割目的序列的效果请参考图3。
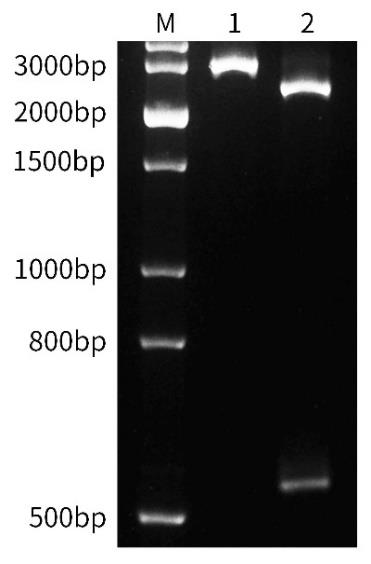
图3.两步法sgRNA合成试剂盒合成的sgRNA引导Cas9核酸酶切割目的基因的效果图。设置27μL反应体系,其中包含1×Cas9 Reaction Buffer,30nM sgRNA,30nM Cas9 Nuclease,25℃孵育10min使sgRNA与Cas9 Nuclease充分结合。随后加入3μL底物DNA (终浓度3nM),混合均匀,37℃孵育15min进行切割反应。随后加入1μL 蛋白酶K溶液(20mg/mL),混合均匀,室温孵育10min以降解反应体系中的蛋白酶。取出10μL反应液,加入2μL 6×DNA上样缓冲液进行琼脂糖凝胶(1.5%)电泳,电泳结束后使用NadRed红色核酸染料室温染色15min,并使用凝胶成像系统观察实验结果。泳道1:反应体系中没有加入sgRNA,DNA片段大小为2773bp;泳道2:反应体系中加入了sgRNA,切割产生的DNA片段大小分别为2206bp和567bp。如图所示,使用本试剂盒合成的sgRNA可以有效引导Cas9 Nuclease充分切割目的DNA。不同实验条件产生的效果可能有所不同,图中效果仅供参考。

- 靶DNA序列的选择和PCR上游引物(即Target-specific DNA oligo)的设计与合成:
- 靶DNA序列的选择。选择的靶DNA序列需要满足如下2个条件。以pUCm-T载体作为基因编辑的靶基因为例展示的对于靶DNA序列的选择请参见图4。
- 选择的靶DNA序列的3'端必须紧邻着PAM序列(NGG)。只有PAM序列上游的20nt DNA序列才能被选择用于CRISPR/Cas9系统的靶序列。
- 任何紧邻着PAM序列(NGG)的靶序列都可被选用。但为了降低切割时的脱靶概率,整个靶序列(包括PAM序列)与其它任何靶DNA序列之外的基因组DNA序列至少有三个错配碱基。如果错配碱基出现在PAM序列或紧邻着PAM序列,会降低脱靶效应的几率。
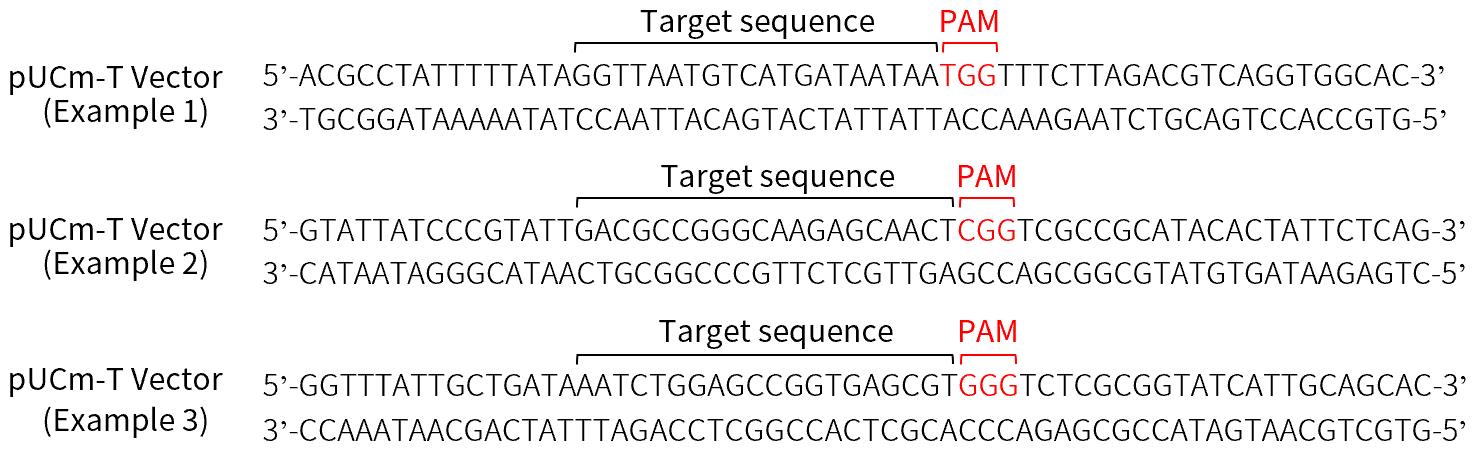
图4.基于CRISPR/Cas9系统的基因编辑靶DNA序列的选择示例。以pUCm-T载体作为基因编辑的靶基因为例展示对于靶DNA序列的选择。
- 选择的靶DNA序列的3'端必须紧邻着PAM序列(NGG)。只有PAM序列上游的20nt DNA序列才能被选择用于CRISPR/Cas9系统的靶序列。
- PCR上游引物(即Target-specific DNA oligo)的设计与合成。PCR上游引物必须包括如下的4部分序。
- 引物的5'端为T7启动子序列(17nt)加额外的四个保护碱基(4nt)。
- 转录起始位点0~2个G。添加的G的个数依赖于靶序列的5'端。T7启动子至少需要两个G才能有效转录。如果选择的靶序列已经包含2个G,就不需要在转录起始位点添加额外的G。如果包含1个或0个G,就需要相应增加1个或2个额外的G。超过2个G会降低切割效率。
- 20nt长度的sgRNA靶序列。
- 引物3'端14nt长度的Scaffold Template退火序列。以pUCm-T载体作为基因编辑的靶基因为例展示的PCR上游引物设计请参见图5。
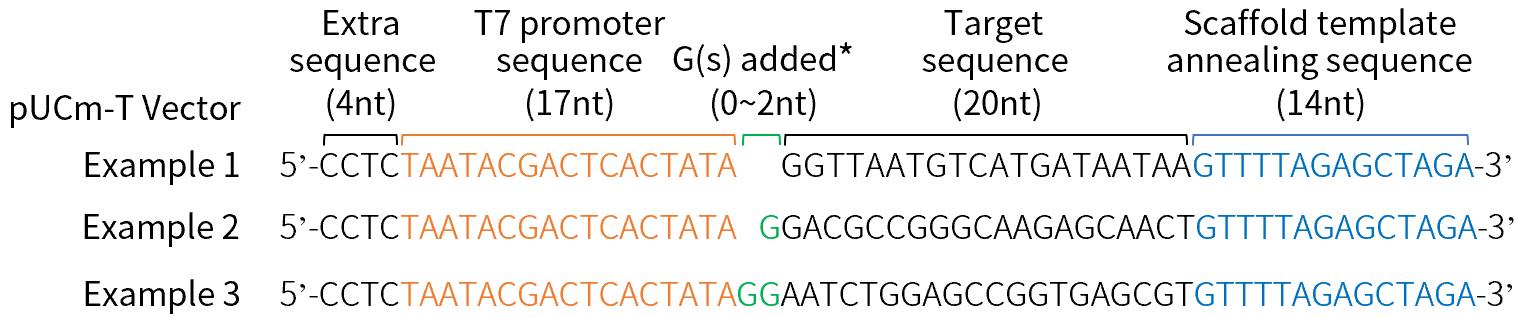
图5.两步法sgRNA合成试剂盒所需PCR上游引物的设计示例。以pUCm-T载体作为基因编辑的靶基因为例展示设计的PCR上游引物序列。
- 引物的5'端为T7启动子序列(17nt)加额外的四个保护碱基(4nt)。
- 靶DNA序列的选择。选择的靶DNA序列需要满足如下2个条件。以pUCm-T载体作为基因编辑的靶基因为例展示的对于靶DNA序列的选择请参见图4。
- PCR扩增合成sgRNA转录所需DNA模板:
- 溶解PCR反应所需的各种溶液。
- 参考下表在冰浴上配制PCR反应体系。如果有多个类似的PCR反应,可以先把下表中除了自行设计合成的Target-specific DNA Oligo之外的所有组分提前预混合,然后分装到各PCR反应管内,再在各管内加入Target-specific DNA Oligo。
成分 用量 2×Template Mix 10μL Target-specific DNA Oligo(10μM) 1μL 超纯水 8.5μL BalbFusion DNA聚合酶 0.5μL 总体积 20μL - 由于此步骤产生的PCR产物后续要作为模板产生sgRNA,所以需要严格按照RNA操作的规范进行,避免RNase污染,相关试剂和耗材需要确保是RNase free的,或者是经过DEPC处理已经去除RNase的。
- 用移液器轻轻吹打混匀或轻微Vortex混匀,室温离心数秒,使液体积聚于管底。
- 如果PCR仪没有热盖,则在PCR管内滴入一滴矿物油。
- 将设置好的PCR反应管置于PCR仪上,开始PCR反应。PCR反应参数的设置可以参考如下表格。
PCR步骤 温度 时间 循环数 步骤1(初始变性) 92℃ 3min 1 步骤2(变性) 92℃ 30s 30 步骤3(退火) 55℃ 30s 步骤4(延伸) 68℃ 30s 步骤5(最终延伸) 68℃ 10min 1 步骤6(最终延伸) 4℃ forever - - PCR反应结束后,可以取约3~5μL PCR产物用于2%或3%的DNA琼脂糖凝胶电泳分析。检测PCR扩增产物条带的单一性和大致产量。如果条带比较单一,产量也符合预期(预期20μL PCR产物中约含有8μg模板DNA),就可以进行后续步骤了。如果产量略偏低,后续的sgRNA的产量也会相应下降。
- 溶解PCR反应所需的各种溶液。
- 体外转录获得sgRNA:
- 溶解并混匀转录反应所需的各种溶液,将T7 RNA聚合酶置于冰浴上或冰盒内。
- 参考下表在冰浴上配制如下转录反应体系。如果同时进行多个转录反应,可以把下表中除sgRNA Template之外的所有组分提前预混合,然后分装到各反应管内,再在各管内加入sgRNA Template。
成分 用量 2×sgRNA Synthesis Buffer 10μL sgRNA模板(PCR产物) 5μL RNase抑制剂(40U/μL) 1μL T7 RNA聚合酶 1μL 超纯水 3μL 总体积 20μL - 由于涉及RNA操作,需要严格按照RNA操作的规范进行,避免RNase污染,相关试剂和耗材需要确保是RNase free的,或者是经过DEPC处理已经去除RNase的。可以适当通过按比例放大反应体系,以获得更多的sgRNA产量。放大反应体系对sgRNA的得率基本没有影响。
- 按照上表配制好反应体系后,轻轻混匀(可以用移液器吹打混匀或用Vortex在最低速度轻轻混匀),瞬时离心。
- 反应条件:37℃孵育0.5~4h。通常反应时间越长,生成的sgRNA越多。
- 终止反应:70℃孵育10min以终止反应。
- 溶解并混匀转录反应所需的各种溶液,将T7 RNA聚合酶置于冰浴上或冰盒内。
- sgRNA的纯化:
- 取20μL转录反应产物,加入2μL DNase I,37℃孵育15min以充分降解DNA模板。
- 后续推荐使用如下的酚氯仿抽和乙醇沉淀方法进行sgRNA的纯化。也可以使用柱式动物小RNA提取试剂盒进行sgRNA的纯化,但该试剂盒每个纯化柱的抽提容量相对较小一点,不能满足10~40μg sgRNA的纯化要求,但可以用于少量sgRNA快速纯化和分析鉴定。
- 在上述反应体系中加入160μL超纯水,再加入20μL 3M NaAc,pH5.2 (约200μL最终体积的1/10体积),混匀。
- 加入200μL水饱和酚/氯仿混合溶液(等体积),充分Vortex混匀以变性蛋白。
- 4℃ 12000~14000g离心10min。收集上层液体至新的1.5mL离心管(Nuclease free),加入2倍体积无水乙醇,混匀,-20℃沉淀3h或-80℃沉淀1h (如果想得到更高产量的sgRNA,可以考虑沉淀过夜)。
- 4℃ 12000~14000g离心10min。去除上清,加入1mL 70%乙醇。
- 4℃ 12000~14000g离心5min。去除上清,尽量洗净残留液体,打开管盖,室温放置2~5min挥发残留乙醇。
- 加入20μL超纯水溶解沉淀,-80℃冻存。sgRNA的浓度可以通过测定紫外吸收,以及通过电泳染色进行确定。通常一个反应体系可以获得约10~40μg sgRNA。
- 取20μL转录反应产物,加入2μL DNase I,37℃孵育15min以充分降解DNA模板。
- (选做)所设计的sgRNA的效果鉴定:
- 参考下表在冰浴上配制如下反应体系。Cas9 Nuclease可以向百奥莱博购买。
成分 用量 超纯水 20μL 10×Cas9 Reaction Buffer 3μL 1μM Cas9 Nuclease 1μL 300nM sgRNA 3μL(终浓度30nM) 25℃预孵育10min 30nM底物DNA 3μL(终浓度3nM) 总体积 30μL - 反应条件:37℃孵育15min。反应时间也可以根据具体的实验目的适当延长至例如30~120min。
- 终止反应:加入1μL 蛋白酶K溶液(20mg/mL),混匀,室温孵育10min。
- 电泳检测:取出10μL反应液,加入2μL 6×DNA上样缓冲液(货号:YT418)进行琼脂糖凝胶电泳,电泳结束后使用NadRed红色核酸染料室温染色15min,并使用凝胶成像系统观察实验结果。
- 结果分析:对于设计的不同的sgRNA,可以根据电泳结果中对于底物DNA的切割效率判定sgRNA的设计效果。
- 参考下表在冰浴上配制如下反应体系。Cas9 Nuclease可以向百奥莱博购买。

- sgRNA获得量低。
- 模板DNA质量问题:DNA中残留的乙醇、酚、氯仿等有机溶剂会影响转录效率。解决方法:重新制备高纯度的DNA模板。
- RNA酶污染:RNA酶可以由模板DNA或者操作中带入。RNA酶会降解转录产物,从而使获得的sgRNA量减少。
- 模板DNA量不足:每个sgRNA转录体系需要约2μg DNA模板。转录前需要定量DNA模板并且电泳检测是否条带单一、清晰。使用更大量的DNA模板对于提高sgRNA产量有一定帮助。
- 模板DNA质量问题:DNA中残留的乙醇、酚、氯仿等有机溶剂会影响转录效率。解决方法:重新制备高纯度的DNA模板。
- sgRNA电泳条带模糊、两条带、大小不正确。sgRNA为100nt左右的单链RNA。在水溶液中容易形成二级结构。因此电泳时位置会发生变化。采用变性后快速冷却处理或者甲酰胺变性凝胶电泳可以显著改善电泳效果。
- sgRNA变性琼脂糖凝胶电泳出现显著小于100nt的弥散带。RNA酶污染造成sgRNA降解。解决方法:采用高质量的无核酸酶酶污染的耗材进行实验。实验过程中注意防止核酸酶污染。
相关搜索:两步法sgRNA合成试剂盒,sgRNA合成,体外转录,体外合成,BalbCRISPR Two-Step sgRNA Synthesis Kit



